¿Cuál es el papel de la cristalografía en la lucha contra el Covid-19?
El trabajo realizado en tiempo record, por la comunidad científica internacional, para resolver la estructura del virus causante del COVID-19 ha sido espectacular, en muy poco tiempo se ha conseguido identificar las posibles dianas para obtener un tratamiento efectivo y más adelante, la vacuna para controlar la pandemia. El papel de la Cristalografía y las Ciencias Estructurales ha sido fundamental. Así nos lo describe la Vicepresidenta de la Asociación Francesa de Cristalografía: Joanna Timmins , en este artículo publicado en la web de la ECA y traducido al español por Juanma García Ruiz.
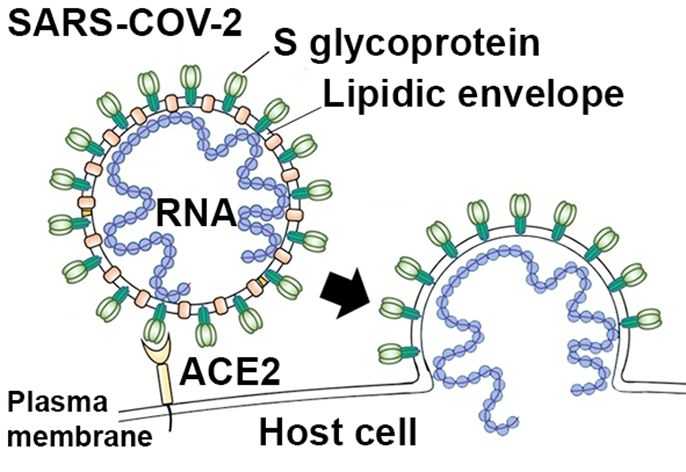
El virus SARS-COV-2 responsable de la actual pandemia de Covid-19, que se está extendiendo progresivamente en todo el mundo, pertenece a la familia de los coronavirus, cuyos primeros miembros fueron identificados hace casi un siglo [1]. Estos virus encapsulados (paquetes de ácidos nucleicos) poseen un genoma de ARN (ácido ribonucleico) y proyecciones o púas superficiales características, reconocibles por microscopía electrónica que les dan un aspecto de corona (de ahí su nombre) (Fig. 1). Estas proyecciones corresponden a una glucoproteína de púas superficial, también conocida como la proteína S, que desempeña un papel clave en la entrada del virus en las células huésped.
Desde hace varias semanas, la comunidad médica y científica de todo el mundo se ha comprometido firmemente a mejorar nuestra comprensión de los detalles de este nuevo virus, el SARS-COV-2, y a encontrar nuevas estrategias terapéuticas y protectoras para poner fin a esta pandemia. Entre estos investigadores, los biólogos estructurales han estado desempeñando un papel esencial en la determinación de las estructuras a escala atómica de las principales proteínas del SARS-COV-2 mediante cristalografía de rayos X y/o criomicroscopía electrónica. Sus estudios estructurales se centran principalmente en dos proteínas virales clave: la glucoproteína S y una proteasa (una enzima capaz de cortar otras proteínas) esenciales para el virus, responsables de la producción de varias proteínas virales a partir de una sola poliproteína.
En la superficie del virus, la glucoproteína S permite que el virus ingrese a las células humanas a través de su interacción con un receptor, identificado como la enzima ACE2, presente en la superficie de las células infectadas [2]. Durante el mes de marzo de 2020, varias estructuras parciales de la proteína SARS-COV-2 S, solas o en complejo con su receptor ACE2, fueron determinadas por cryo-EM, revelando así su modo de reconocimiento del receptor y los mecanismos moleculares subyacentes a la fusión. de la envoltura viral con la membrana plasmática de las células infectadas [2,3,4]. Esta información estructural ahora puede explotarse en primer lugar para identificar inhibidores de estas etapas clave del ciclo viral, que en última instancia podrían servir como medicamentos antivirales y, en segundo lugar, desarrollar vacunas. Al estar en la superficie de estos virus, la glucoproteína S desempeña un papel esencial en la respuesta inmune del huésped. La mayoría de los anticuerpos neutralizantes reconocen esta proteína. El desarrollo de una vacuna eficaz contra el SARS-COV-2 requerirá identificar las regiones de la proteína S que muestran el potencial inmunogénico más fuerte y capaces de inducir una respuesta inmune efectiva y específica contra el virus.
Una segunda proteína viral, objeto de numerosos estudios estructurales, es la principal proteasa del SARS-COV-2, llamada MPRO o 3CLPRO, que actualmente representa uno de los objetivos terapéuticos más prometedores para el desarrollo de medicamentos antivirales contra el SARS-COV-2. El primer paso fue la determinación de la estructura cristalina de esta enzima en alta resolución, que se logró en marzo de 2020 [5,6]. Numerosos equipos de investigación están utilizando estos datos estructurales para identificar inhibidores específicos y efectivos de esta proteasa mediante enfoques teóricos (modelado, diseño de fármacos in silico, etc.) [6] y/o experimentales, en particular determinando las estructuras cristalinas de los complejos entre la proteasa y las pequeñas moléculas inhibidoras. En este contexto, el equipo de XChem ha recopilado grandes cantidades de datos cristalográficos en el Diamond Light Source (uno de los sincrotrones que usan los cristalógrafos) en el Reino Unido, haciendo un barrido masivo de moléculas pequeñas, y recientemente se ha lanzado una convocatoria de voluntarios para encontrar cristalógrafos, biólogos estructurales y químicos que deseen participar en el análisis de estos datos, en el diseño de nuevas moléculas o en la síntesis de inhibidores.
En Francia, siguiendo las medidas del gobierno francés, los principales instrumentos importantes, incluido la Fuente Europea de Radiación Sincrotrón (ESRF), han estado cerrados desde el 17 de marzo de 2020, pero aun así participan en la lucha contra Covid-19. El ESRF, por ejemplo, está listo para proporcionar acceso excepcional a sus instalaciones para proyectos relacionados con Covid-19.
Todos aquellos investigadores que deseen utilizar su experiencia para combatir Covid-19, pueden visitar el sitio: https://crowdfightcovid19.org/.
Los investigadores de CRCA-CBI en Toulouse han establecido esta plataforma para poner las habilidades y la experiencia de la comunidad científica más amplia que actualmente no se dedica a la investigación de Covid-19 al servicio de aquellos que ya están muy involucrados en esta lucha.
Referencias:
- Peiris J. Coronaviruses. Clinical Virology, Fourth Edition. p. 1243-1265.
- Walls AC, Park YJ, Tortorici MA, Wall A, McGuire AT, Veesler D. Structure, Function, and Antigenicity of the SARS-CoV-2 Spike Glycoprotein. Cell 2020 Mar 6. pii: S0092-8674(20)30262-2.
- Yan R, Zhang Y, Li Y, Xia L, Guo Y, Zhou Q. Structural basis for the recognition of SARS-CoV-2 by full-length human ACE2. 2020 Mar 27. 367 (6485) : 1444-1448.
- Wrapp D, Wang N, Corbett KS, Goldsmith JA, Hsieh CL, Abiona O, Graham BS, McLellan JS. Cryo-EM structure of the 2019-nCoV spike in the prefusion conformation. 2020 Mar 13. 367 (6483): 1260-1263.
- Zhang L, Lin D, Sun X, Curth U, Drosten C, Sauerhering L, Becker S, Rox K, Hilgenfeld R. Crystal structure of SARS-CoV-2 main protease provides a basis for design of improved α-ketoamide inhibitors. 2020 Mar 20. pii : eabb3405.
- Coronavirus SARS-CoV2: BESSY II data accelerate drug development. 2020 March 19.
- Chen YW, Yiu CB, Wong KY. Prediction of the SARS-CoV-2 (2019-nCoV) 3C-like protease (3CL (pro)) structure: virtual screening reveals velpatasvir, ledipasvir, and other drug repurposing candidates. 2020 Feb 21. 9:129.
Joanna Timmins, es vicepresidenta de la Asociación Francesa de Cristalografía
Artículo Publicado el 8 de abril de 2020. Versión original en francés: 3 de abril de 2020
Traducción libre de Juanma García-Ruiz
Compartir:



